1. EINFÜHRUNG
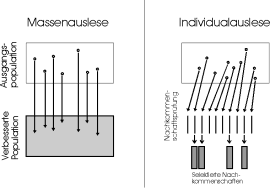
1.1. Massenauslese
1.2. Individualauslese mit
Nachkommenschaftsprüfung
2. AUSLESEZÜCHTUNG
3. SELEKTION BEI SELBSTBEFRUCHTERN (in der Kreuzungszüchtung)
3.1. Bulk - Methode
3.2. Pedigree - Methode
3.3. Andere Methoden
3.4. Selektion zwischen
Kreuzungen
4. EINFACHE SELEKTIONSVERFAHREN
4.1. Mindestleistungsselektion
4.2. Tandemselektion
5. MULTIVARIATE SELEKTIONSVERFAHREN
5.1. Sortenwertindex
5.2. Abstandsmaßindex
oder "Selektion nach Ähnlichkeit"
6. LITERATUR
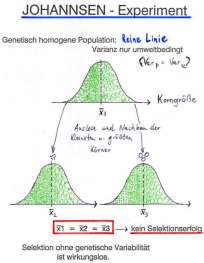
Ständige Selektion innerhalb eines vorhandenen Genotypenspektrums einerseits und andererseits Prozesse, die andauernd neue genetische Variabilität hervorbringen (Rekombination u. Mutation), sind grundlegende Vorgänge, die der Evolution der Arten und der landwirtschaftlichen Pflanzenzüchtung gemeinsam sind. Während jedoch der Effekt der natürlichen Selektion in der Evolution stets in einer Verbesserung der reproduktiven Fitneß unter spezifischen Umweltbedingungen besteht (Anpassung), sind die Selektionsziele in der Pflanzenzüchtung vom Menschen vorgegebene und veränderliche Anforderungen unterschiedlichster Art. Seit vermutlich mehr als zehn Jahrtausenden nutzt der Mensch die natürliche Formenmannigfaltigkeit der Arten, indem er Selektion betreibt; vor etwa 100 Jahren begann er, auch die genetische Variabilität selbst auf systematische Weise zu erhöhen, um damit letztlich seine Selektionsmöglichkeiten entscheidend zu verbessern, da Selektion nur erfolgreich sein kann, wo eine genügend große genetische Variabilität (erblich bedingte Unterschiedlichkeit zwischen Individuen) vorliegt - Selektion ohne vorhandene Variabilität bleibt wirkungslos (vgl. die Selektionsexperimente von JOHANNSEN, 1903).

Aus dem Gesagten ergeben sich zwei Problemkreise für die praktische Pflanzenzüchtung:
1. Das Problem des Erkennens brauchbarer Genotypen: Selektion
nach leicht erkennbaren, oft monogen vererbten Merkmalen ist relativ einfach
durchzuführen. Um aber auf polygen bedingte, quantitative Eigenschaften
(die überwiegende Zahl der agronomisch bedeutsamen Merkmale gehört
in diese Gruppe) selektieren zu können, ist es nötig, objektive
Methoden anzuwenden und modifizierende Umwelteinflüsse auszuschalten,
um die brauchbaren Genotypen erkennen zu können.
2. Das Problem der großen Individuenzahl, aus der brauchbare
Genotypen auszulesen sind: Mit der Größe der Population
steigt die Wahrscheinlichkeit, brauchbare Individuen aus derselben auszulesen.
In der Praxis jedoch sind der Größe der Population in den meisten
Fällen sehr enge Grenzen gesetzt (Arbeitskapazität, Bodenfläche,
versch. technische Begrenzungen).
Wichtige B E G R I F F E :
Stabilisierende Selektion:
Individuen mit extremen, stark vom Populationsmittel abweichenden Merkmalsausprägungen
werden aus der Population entfernt, das Populationsmittel wird dadurch
nicht verändert, die Variationsbreite wird jedoch geringer.
Disruptive Selektion:
Extremtypen der Merkmalsverteilung werden selektiert und getrennt weitergeführt,
wodurch nach mehreren Zyklen zwei Untergruppen der Population mit unterschiedlichen
Mittelwerten entstehen.
Beispiel: Aus amerikanischen Maisstämmen mit einem Ausgangsölgehalt
von 4,7 % wurde 50 Jahre hindurch auf Formen mit höherem oder niedrigerem
Ölgehalt (= disruptiv) selektiert, wodurch Stämme mit extremen
Gehalten von 15.4 bzw. 1.0 % erreicht wurden. Auch der Proteingehalt konnte
von anfänglich 12 % nach 90 Generationen Selektion auf bis zu 30 %
erhöht oder auf unter 4 % gesenkt werden.
Gerichtete (lineare) Selektion:
Es werden Pflanzen mit ganz bestimmter Merkmalsausprägung ausgelesen,
wodurch sich das Populationsmittel durch fortgesetzte Selektion kontinuierlich
in Richtung eines bestimmten Zuchtzieles verschieben kann.
Aus einem Pflanzenbestand werden alle jene Pflanzen, die in ihren Eigenschaften
dem Zuchtziel entsprechen, ausgelesen und getrennt vom Rest des Bestandes
gemeinsam (als Ramsch) weitervermehrt (= positive Massenauslese). Oder:
Es werden alle jene Pflanzen, die unerwünschte Merkmalsausprägungen
zeigen, aus der Population entfernt und somit von der weiteren Vermehrung
ausgeschlossen (= negative Massenauslese).
Bei Fremdbefruchtern ist dabei stets eine Isolation der ausgelesenen
Typen notwendig, da eine unkontrollierte Bestäubung den Selektionseffekt
wieder aufhebt (die Effizienz der Selektion hängt bei FB z.B. auch
davon ab, ob die Auslese vor oder erst nach der Blüte durchgeführt
werden kann; die Auslese vor der Blüte verhindert eine Bestäubung
der Population durch unerwünschte Genotypen).
Massenauslese dient heute vor allem der Erhaltung von fertigen
Sorten (eine bestimmte Anzahl von typischen (= dem Sortenbild entsprechenden)
Pflanzen wird selektiert, in Reihen angebaut und beurteilt, abweichende
Reihen werden eliminiert, die verbliebenen werden geerntet und als Ramsch
weitergeführt; Erhaltungszüchtung), kann aber bei der züchterischen
Bearbeitung von Landsorten, die in sich einen hohen Grad an genetischer
Variabilität aufweisen, sehr rasch zu Selektionserfolgen führen
(Auslesezüchtung).
Eine verfeinerte Massenauslese besteht darin, daß die selektierten
Pflanzen getrennt voneinander geprüft (Nachkommenschaftsprüfung
!) und danach die besten Linien wiederum geramscht werden, wodurch eine
Verbesserung der Population zustandekommt.
1.2. Individualauslese mit Nachkommenschaftsprüfung
Aus einem Bestand verschiedener Genotypen werden Einzelpflanzen (bei Getreide zur Einschränkung der Vermischungsgefahr auch Einzelähren) gezogen, deren Nachkommenschaften voneinander getrennt geprüft werden; die eigentliche Selektion erfolgt erst an den Nachkommenschaften, wodurch - im Gegensatz zur Massenauslese, die nur auf dem Phänotyp der jeweils ausgelesenen Pflanzen basiert - der Genotyp (= "Erbwert") der selektierten Pflanzen, der sich eben in den Nachkommenschaften äußert, als Kriterium für die Selektion herangezogen werden kann. Die Nachkommenschaftsprüfung stellt somit eine Art "Erfolgskontrolle" bezüglich der Effektivität eines Selektionsschrittes dar.
Massenauslese und Individualauslese sind in Abb. 1 vergleichend dargestellt.
Die Auslesezüchtung als eigenständige Zuchtmethode kann überall
dort sinnvoll eingesetzt werden, wo in einer Landsorte oder auch Wildpopulation
eine für den Ausleseerfolg genügend große genetische Variabilität
vorhanden ist. In solchen Fällen führt die Auslesezüchtung
(als Massen- oder Individualauslese durchgeführt), die also eine bereits
vorhandene Variation nutzt, zu einer Verbesserung der Ausgangspopulation.
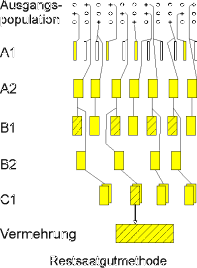
Auslesezüchtung bei Selbstbestäubern und Klonen: Kontinuierliche
Selektion in jeder Generation.
Auslesezüchtung bei Fremdbestäubern: Restsaatgutmethode,
um Bestäubung durch unerwünschte Polleneltern zu verhindern.
3. SELEKTION BEI SELBSTBEFRUCHTERN (in
der Kreuzungszüchtung)
3.1. Bulk - oder Ramsch - Methode
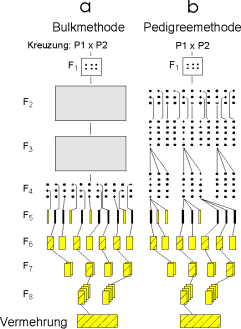
Die Bulk- oder Ramschmethode zur Behandlung spaltender Selbstbefruchtergenerationen reproduziert Kreuzungsprodukte von F1 bis F5 od. F6 ohne Selektion; dabei werden nach einer Kreuzung in den Spaltungsgenerationen möglichst viele Genotypen vermehrt, individuelle Selektion (mit Nachkommenschaftsprüfung) setzt hingegen erst ein, wenn die Kreuzungspopulation einen hohen Homozygotiegrad erreicht hat und nur mehr wenig Aufspaltung zeigt.
Vorteile der Bulk-Methode gegenüber anderen Verfahren:
1. Da die sehr aufwendige Selektion in spaltenden Generationen nicht
vorgenommen werden muß, kann eine größere Anzahl an Kreuzungen
arbeitsmäßig bewältigt werden.
2. Natürlicher od. künstlicher Selektionsdruck kann eine
große Anzahl an unerwünschten Typen eliminieren (z.B. wenn photoperiodische
Anpassung, Krankheitsresistenz oder Frosttoleranz gefordert werden).
3. Es entfällt die Frage, ob und inwiefern brauchbare Genotypen
im heterozygoten Zustand erkannt und daher sinnvoll selektiert werden können.
Nachteile der Bulk-Methode:
1. Durch natürliche Selektion können auch die für die
Erreichung des Zuchtzieles brauchbaren Typen eliminiert werden.
2. Durch Konkurrenz innerhalb von spaltenden Populationen können
ebenfalls brauchbare Genotypen verloren werden (kleinsamige Typen z.B.
können sich in einem Ramsch durchsetzen, weil solche Genotypen zumeist
einen höheren Vermehrungskoeffizienten aufweisen; spätreifende
Genotypen können z.B. bei der Sojabohne frühreifende verdrängen).
In Abb. 2a ist der Verlauf eines Bulk - Verfahrens schematisch dargestellt.
3.2. Pedigree - oder Stammbaum - Methode
Die Pedigree- oder Stammbaummethode zeichnet sich im Vergleich zur Bulkmethode dadurch aus, daß bereits in den ersten Spaltungsgenerationen nach einer Kreuzung (etwa in F2 od. F3) mit der Selektion begonnen wird, die systematisch bis zum Erreichen der Homozygotie fortgesetzt wird. Selektion erfolgt daher zunächst an heterozygoten Individuen, nach einigen Generationen werden Familienmerkmale sichtbar, innerhalb der Familien (Einzelpflanzennachkommenschaften) kommt es jedoch noch zu Aufspaltung, sodaß nun (F3 od. F4) nach den besten Einzelpflanzen innerhalb von Familien selektiert wird. Später ist eine Selektion innerhalb von Familien nicht mehr effektiv (durch die erreichte Homozygotie), weshalb dann eine Selektion zwischen den Familien erfolgt. Etwa ab F6 od. F7 ist es möglich, in hohem Maße homozygote Individuen auszulesen, die sich genetisch konstant verhalten und als Stämme weiteren Prüfungen unterzogen werden können.
Im Hinblick auf die unterschiedliche Heritabilität einzelner Merkmale
muß eine gewisse Selektionssequenz in den Pedigreegenerationen eingehalten
werden: Auf Eigenschaften mit hoher Heritabilität (Merkmalsausprägung
an einzelnen oder wenigen Pflanzen relativ sicher erkennbar) kann in frühen
Generationen selektiert werden, wodurch die Kreuzungspopulation auf bestimmte,
dem Zuchtziel entsprechende Typen eingeengt wird (brauchbare Genotypen
werden konzentriert); dagegen erlauben Eigenschaften, die eine eher niedrige
Heritabilität aufweisen, eine Selektion erst später, wenn wiederum
Homozygotie erreicht ist, und wenn wiederholte Prüfung des Materials
(ev. mehrortig u. mehrjährig) möglich wird. Dadurch ergeben sich
etwa für die selbstbefruchtenden Getreidearten folgende Selektionsmöglichkeiten
(Tabelle):
| Heritabilität | Merkmal | Selektionsbeginn |
| hoch | Best. Resistenzeigenschaften, Reifezeit, Begrannung, Kornfarbe, Spelzenform | F2 - F3 |
| mittel | Korn- u. Ährenmerkmale, Standfestigkeit, Qualitätsmerkmale, ggf. Halmlänge | F4 - F5 |
| niedrig | Ertragskomponenten, Ertragspotential, versch. physiol. Eigenschaften | F7 - F8 |
Abb. 2b zeigt schematisch ein typisches Pedigreeverfahren.

Für die Züchtung von Liniensorten bei Selbstbestäubern ist die Pedigreemethode gegenwärtig die am öftesten angewandte Züchtungsmethode. Zahlreiche Modifikationen der Methode hängen unter anderem von der Kulturart, dem genetischen Material (Kreuzungskombination, Populationsgröße etc.) und von den zu selektierenden Merkmalen ab.
Methode der Teilpopulationen:
Die Methode der Teilpopulationen (Teilramschmethode, Methode der Familienzüchtung,
Bulk-Progeny-Methode) stellt eine Kombination von Stammbaum- und Ramschmethode
dar. Dabei wird eine große Zahl an F2- od. F3-Pflanzen ausgelesen,
deren Nachkommenschaften als getrennte Ramsche vermehrt und geprüft
werden. Schließlich werden nach dem Erreichen der Homozygotie aus
den besten dieser Teilramsche Einzelpflanzen gezogen und als Linien/Stämme
weiter bearbeitet (geprüft).
Als Vorteil der Teilramschmethode gegenüber anderen Verfahren
wird die Möglichkeit angesehen, bereits in F4 od. F5 mehrortig prüfen
zu können, was eine Selektion auf Eigenschaften mit geringer Heritabilität
mittelbar in relativ frühen Generationen bedeutet.
Methoden, die eine Beschleunigung oder Verkürzung des Züchtungsganges zum Ziel haben:
Single-Seed-Descent - Methode:
Zur Durchführung der SSD- od. Einkornramschmethode wird eine größere Zahl an F2-Samen angebaut und von jeder resultierenden Pflanze zumindest ein Same weitergeführt; dies kann im Glashaus auf kleinem Raum mit mehreren Generationen pro Jahr erfolgen, sodaß in relativ kurzer Zeit homozygote Pflanzen aus einer Kreuzung zur Verfügung stehen, an deren Nachkommenschaften dann in verschiedenen Stufen die SELEKTION durchgeführt wird; die spaltenden Generationen werden somit gänzlich ohne Selektion und vergleichsweise rasch überwunden (das gilt besonders für sommerannuelle Kulturarten mit sehr geringem Vernalisationsbedarf wie z.B. Sommergerste od. Sojabohne), andererseits kann aber (ähnlich wie bei der nachfolgend dargestellten Haploidenmethode) nie das gesamte "Rekombinationspotential" einer Kreuzung ausgeschöpft werden (Aufspaltungen zwischen den Nachkommenschaften der einzelnen F2-Pflanzen werden im Falle von SSD nicht genutzt). Die SSD-Methode kommt in verschiedenen Modifikationen gegenwärtig vor allem in der Sojabohnenzüchtung zum Einsatz, wo weit mehr als die Hälfte der aktuellen Sorten aus Single-Seed-Descents hervorgeht.
Haploiden - Methode:
Die Haploiden- od. Antherenkultur-methode geht von der Möglichkeit aus, mit Hilfe von Gewebekulturtechniken aus Antheren oder isolierten Pollenkörnern von heterozygoten Pflanzen (etwa F1- od. F2-Generation) haploide Pflanzen zu gewinnen, die nach ihrer Diploidisierung (spontan oder mittels Mitosehemmer wie z.B. Colchizin oder Oryzalin) vollkommen homozygote Pflanzen (sog. Dihaploide) ergeben, die als reine Linien vermehrt, geprüft und der Selektion unterzogen werden können. Dieses Verfahren, das eine enorme Zeitersparnis in der Linienzüchtung bei Selbstbefruchtern bedeuten würde, bedarf gegenwärtig bei vielen Arten noch gewisser Verbesserungen, ehe es in die praktische Züchtung breiten Eingang finden kann. Bei Gerste, Reis, Raps, versch. anderen Cruciferen, Solanaceae etc. wird es jedoch bereits erfolgreich zur praktischen Sortenentwicklung eingesetzt.
3.4. Selektion zwischen Kreuzungen
Neben der Selektion innerhalb der Nachkommenschaften einer Kreuzung
ist auch die Selektion zwischen Kreuzungskombinationen (und zuvor auch
die Wahl von Kreuzungseltern) ein bedeutender Aspekt der Selektion innerhalb
eines Zuchtprogrammes. Kreuzungen werden aus unterschiedlichen züchterischen
Intentionen durchgeführt, Kreuzungskombinationen unterscheiden sich
im Populationsmittelwert und in der genotypischen Varianz. Der Populationsmittelwert
der Kreuzung hängt von den Leistungswerten der beiden Eltern im betreffenden
Merkmal ab, wogegen die Varianz der Kreuzung von der genetischen Distanz
der Eltern bestimmt wird und umso größer ist, je stärker
sich die Eltern genetisch unterscheiden. Kreuzungen werden praktisch meist
visuell beurteilt und unterschiedlich behandelt (Selektionsbeginn, Selektionsintensität,
Populationsgröße etc.), können aber auch durch Prüfung
von Kreuzungsramschen etwa in der F3 miteinander verglichen und damit selektiert
werden.
4. EINFACHE SELEKTIONSVERFAHREN
4.1. Mindestleistungsselektion
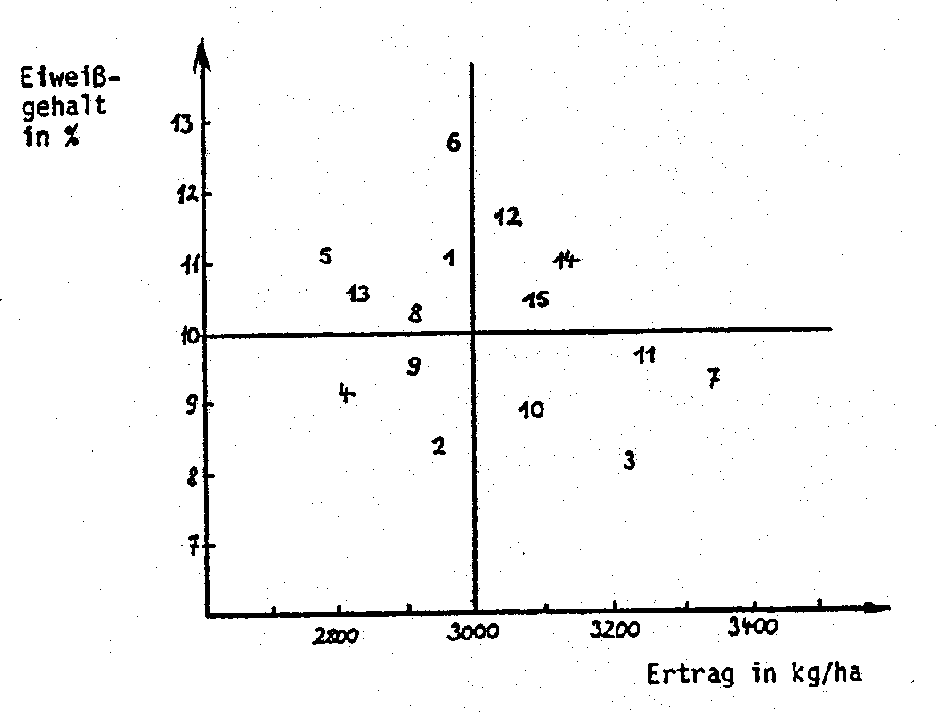
Die Mindestleistungsselektion oder Selektion nach unabhängigen Grenzen ist ein Selektionsverfahren, bei dem in einer Generation gleichzeitig auf mehrere Merkmale selektiert wird, wobei die Selektionsgrenze für jedes Merkmal unabhängig von der Leistungshöhe in anderen Merkmalen festgelegt wird; alle Stämme also, die in einem Merkmal eine bestimmte, vorgegebene Mindestleistung nicht erreichen, werden ohne Berücksichtigung ihrer anderen Eigenschaften eliminiert. Dieses Verfahren schließt somit eine Kompensationsmöglichkeit zwischen einzelnen Merkmalen (Teileigenschaften, Ertragskomponenten) aus, weshalb bei seiner Anwendung die Gefahr besteht, daß wertvolles Material aufgrund einer zu geringen Leistung in einem Merkmal verlorengeht.
Abb. 3 zeigt ein Beispiel für die Selektion nach Mindestleistung in zwei Merkmalen.
Als Tandemselektion wird ein Verfahren bezeichnet, bei welchem in einer Generation auf jeweils ein Merkmal ausgelesen wird. Für eine solche Vorgangsweise ist ein umfangreiches Zuchtmaterial nötig, auch darf die Selektionsintensität in den einzelnen Generationen nicht zu groß sein, was jedoch andererseits den Selektionserfolg beeinträchtigt; auch hier bleibt die Kompensationsmöglichkeit zwischen Teileigenschaften unberücksichtigt, da Selektionsgrenzen unabhängig festgelegt werden.
Eine durchaus sinnvolle Anwendung der Tandemselektion würde bei Einhaltung einer Selektionssequenz analog jener der Pedigree-Methode gegeben sein, wo Merkmale je nach Heritabilität in früheren oder späteren Generationen ausgelesen werden.
5. MULTIVARIATE SELEKTIONSVERFAHREN
Im folgenden werden aus der Vielzahl von multivariaten Selektionsverfahren
zwei relativ einfache Indexmethoden vorgestellt, der Sortenwertindex von
SVAB und der Abstandsmaßindex von SCHWARZBACH.
5.1. Sortenwertindex
Der Sortenwertindex (SVAB, 1976) wurde eigentlich zur Beurteilung des
ökonomischen Gesamtwertes von Industriepflanzen entwickelt, ist aber
in der Züchtung durchaus allgemein einsetzbar. Dabei werden verschiedene
Eigenschaften, die in unterschiedlichen Dimensionen vorliegen, wie etwa
Gewicht, Länge, Resistenz, Standfestigkeit, Qualität etc., in
einem INDEXWERT zusammengefaßt.
Die Methode des Sortenwertindex beruht auf einem multiplikativen Modell,
jede Eigenschaft wird durch einen Wertkoeffizienten wj (Transformationswert
des Meßwertes des j-ten Merkmales) dargestellt. Der Gesamtwert einer
Sorte mit k Merkmalen wäre demnach
Wg = w1 x w2 x w3 .... x wk.
Die Wertkoeffizienten für die einzelnen Merkmale sind die als Prozente eines Zielwertes ausgedrückten Meßwerte, sie können Werte zwischen 0 und 1 annehmen oder sogar >1 sein, wenn der Meßwert für ein Merkmal einer Sorte besser als der Zielwert ist. Für jede Eigenschaft muß also ein sinnvoller Zielwert vorgegeben werden, dem jeweils der Wert-koeffizient 1.0 zugeordnet wird. Der Gesamtwert der sich so ergebenden "Idealsorte I" ist daher
Wg(I) = 1.0 x 1.0 x 1.0 x .... x 1.0 = 1.0 .
Eine hypothetische Sorte A, die im ersten Merkmal um 20 % niedriger, im zweiten um 9 % niedriger, im dritten um 7 % höher als die Idealsorte liegt und in allen anderen Eigenschaften der Idealsorte gleicht, hat daher einen Gesamtwert von
Wg(A) = 0.80 x 0.91 x 1.07 x .... x 1.0 = 0.779 .
Der Gesamtwert der Sorte A beträgt somit rund 78 % gemessen an der Idealsorte. Diese errechneten Gesamtwerte beziehen sich auf eine beliebige Ertragseinheit; durch Multiplikation eines Gesamtwertes mit einem festgestellten Flächenertrag kann auch der Ertrag im Sortenwertindex berücksichtigt werden, für die Sorte A mit einem Ertrag von 20 dt/ha würde sich demnach ein Gesamtwert je Flächeneinheit von
Wg(A) dt/ha = 0.779 x 20 = 15.6 dt/ha
ergeben.
Vergleichbar diesem Sortenwertindex wird teilweise z.B. der Wert von Sojabohnen-Genotypen beurteilt: Dabei wird ein einfacher wirtschaftlicher Index aus Ölgehalt x Sojaöl-Weltmarktpreis und Proteingehalt x Protein-Weltmarktpreis gebildet und mit dem Ertrag multipliziert. Als Selektionskriterium dient also der tatsächliche monetäre Produktwert, der aus unterschiedlichen Genotypen erzielbar ist.
5.2. Abstandsmaßindex oder "Selektion nach Ähnlichkeit"
Die Methode des Abstandsmaßes (SCHWARZBACH, 1972) wurde entwickelt, um auch bei umfangreichem Zuchtmaterial und gleichzeitiger Selektion nach mehreren Merkmalen eine Bewertung vornehmen zu können.
Das Prinzip dieser Methode besteht darin, daß für jedes Merkmal ein ZUCHTZIEL festgelegt wird und dann der ABSTAND jedes zu beurteilenden Zuchtstammes vom Zuchtziel nach dem pythagoräischen Lehrsatz errechnet wird. Dazu ist es zunächst notwendig, die in unterschiedlichen Dimensionen vorliegenden Meßwerte der verschiedenen Merkmale vergleichbar zu machen, was durch Umrechnung der Meßwerte in Standardeinheiten nach der folgenden Formel geschieht:
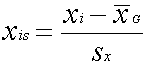
wobei xis der standardisierte Wert, xi der jeweilige Beobachtungswert, xG der Mittelwert aus allen Beobachtungen eines Merkmals und sx die Standardabweichung aller Beobachtungen ist. Nun kann man für die zwei Merkmale ein Zuchtziel vorgeben und den Abstand eines bestimmmten Zuchtstammes vom Zuchtziel mit dem pythagoräischen Lehrsatz ermitteln:
wobei d der Abstand des Stammes vom Zuchtziel ist, az und bz die Zuchtziele
für die beiden Merkmale und ai und bi die Merkmalswerte des Stammes
sind.
Berücksichtigt man gleichzeitig drei Merkmale, so läßt
sich die Ermittlung des Abstandes zwischen Zuchtziel und einem Stamm in
einer dreidimensionalen Graphik darstellen oder nach Erweiterung der obigen
Distanzformel wie folgt berechnen:
Bei Berücksichtigung von mehr als drei Merkmalen liegt der Abstand zwischen Zuchtziel und Stamm in einem mehrdimensionalen Raum und ist daher nicht mehr vorstell bzw. graphisch darstellbar, er kann jedoch mit einer je nach Anzahl der Merkmale erweiterten Distanzformel leicht errechnet werden.
Zur Beurteilung einer großen Zahl von Zuchtstämmen nach dem beschriebenen Verfahren ermittelt man die Entfernungen der Stämme vom Zuchtziel und ordnet dieselben dann nach größer werdendem Abstand zum Zuchtziel tabellarisch, sodaß man eine Rangreihung aller Stämme erhält.
Gewichtung von Merkmalen:
Oft ist es zweckmäßig, das Zuchtziel nicht in realen Merkmalswerten
vorzugeben, sondern als Anzahl an Standardabweichungen über dem jeweiligen
Mittelwert festzulegen, was eine Auslese "in eine bestimmte Richtung" bedeutet.
Dabei ist es auch möglich, für gewisse Merkmale eine größere
Anzahl an Streuungseinheiten vom Mittelwert vorzugeben, was eine intensivere
Selektion auf diese Merkmale und damit eine "Gewichtung" der Eigenschaften
darstellt.
Ein stärkeres Mittel zur Veränderung der Selektionsintensität
von Merkmalen stellt die Möglichkeit dar, eine Gewichtung mit Hilfe
von Faktoren vorzunehmen; dabei werden die quadrierten Merkmalsdifferenzen
mit vom Züchter festgelegten Faktoren multipliziert und die Formel
für das Abstandsmaß ändert sich wie folgt:
Bei einer solchen Art der Gewichtung ist das Verhältnis der Faktoren zueinander von Bedeutung, nicht jedoch deren absolute Größe.
BAKER, R. J., 1986, Selection indices in plant breeding,
CRC Press, Boca Raton, Florida.
BECKER, H., 1993, Pflanzenzüchtung, Verlag Eugen
Ulmer, Stuttgart, UTB 1744.
FISCHBECK, G., W. PLARRE & W. SCHUSTER (Hrsg.), 1985,
Lehrbuch der Züchtung landwirtschaftlicher Kulturpflanzen, Band 2,
spezieller Teil, zweite, neubearb. Auflage, P. Parey, Berlin.
HADDAD, N. I. & F. J. MUEHLBAUER, 1981, Comparison
of random bulk population and single-seed-descent methods for lentil breeding,
Euphytica 30:643-651.
HOFFMANN, W., A. MUDRA & W. PLARRE, 1971, Lehrbuch
der Züchtung landwirtschaftlicher Kulturpflanzen, Band 1, allgemeiner
Teil, P. Parey, Berlin.
JOHANNSEN, W., 1903, Über Erblichkeit in Populationen
und in reinen Linien, Fischer, Jena.
KEMPTON, R. A. & P. N. FOX (eds.), 1997, Statistical
methods for plant variety evaluation, Chapman & Hall, London.
ODENBACH, W. (Hrsg.), 1997, Biologische Grundlagen der
Pflanzenzüchtung, Parey Buchverlag, Berlin.
RUCKENBAUER, P., 1982, Grundlegende Aspekte der Anwendung
von Indizes in der Pflanzenzüchtung, Arbeitstagung, Arbeitsgemeinschaft
Saatzuchtleiter Gumpenstein, 1-14.
SCHMALZ, H., 1980, Pflanzenzüchtung, dritte Auflage,
VEB Deutscher Landwirtschaftsverlag, Berlin.
SCHNELL, F. W. & H. C. BECKER, 1982, Index-Selektion
bei Mais, diskutiert an einer Prüfungsserie mit Experimentalhybriden,
Arbeitstagung, Arbeitsgemeinschaft Saatzuchtleiter Gumpenstein, 157-166.
SCHWARZBACH, E., 1982, Die Dilemmas bei der Zusammenfassung
vieler Merkmale einer Sorte zu einer Zahl, Arbeitstagung, Arbeitsgemeinschaft
Saatzuchtleiter Gumpenstein, 151-155.
SCHWARZBACH, E., 1972, Einige Anwendungsmöglichkeiten
elektronischer Datenverarbeitungsanlagen (EDV) für die Beurteilung
von Zuchtmaterial, Arbeitstagung, Arbeitsgemeinschaft Saatzuchtleiter Gumpenstein,
277-287.
STOSKOPF, N. C. (with D.T. TOMES & B.R. CHRISTIE),
1993, Plant breeding, theory and practice, Westview Press, Boulder.
SVAB, J., 1976, Sortenwertindex, Arbeitstagung, Arbeitsgemeinschaft
Saatzuchtleiter Gumpenstein, 153-156.
Mehr zum Thema Selektion:
Übung "Genetische Diversität"
(Indexselektion, multivariate Selektion)
Übung "Quantitative Genetik"
(Heritabilität und Selektionserfolg)